中国农科院研究员团队、入选国家高层次人才计划,在《Nature》子刊再获重要进展,实现植物调控因子精准捕获
- 2026-02-04 21:49:38
近日,中国农业科学院蔬菜花卉研究所蔬菜分子设计育种创新团队成功开发了一种名为“邻近空间蛋白标记系统”(CSPL)的新型技术,用于高效捕获与植物基因组特定DNA序列结合的调控蛋白,例如启动子区域的转录因子。这一突破性工具为深入解析目标基因上游调控网络提供了强有力的技术支撑,相关研究成果以A CRISPR-based sequence proximity binding protein labelling system for scanning upstream regulatory proteins为题,已发表于国际权威期刊《Nature Plants》。去年,他们还以“Transposon proliferation drives genome architecture and regulatory evolution in wild and domesticated peppers”为题在《Nature Plants》发表了相关研究成果。点击阅读:
继去年Nature Genetics后,中国农科院“国家优青”团队又在Nature Plants发表重要研究成果


该研究源于团队在甘蓝类蔬菜中的长期探索。他们发现,约70%的基因表达变化与结构变异(SV)密切相关,而部分SV区域富集了大量转录因子结合位点。基于此,团队此前已成功定位了甘蓝叶球驯化过程中的关键基因,如ACS和KAN1(发表于《Nature Genetics》,2024),并挖掘出参与叶球发育调控的重要基因PIF4(发表于《Plant Journal》,2022)。然而,这些基因如何被上游信号精确调控,其背后的分子机制仍不清晰。为破解这一难题,研究团队致力于开发一种能够高效鉴定目标基因上游调控因子的新方法。
传统技术如染色质免疫共沉淀(ChIP)在鉴定转录因子等调控蛋白时面临诸多挑战:这些蛋白与DNA的相互作用往往瞬时、微弱且丰度极低,加之依赖特异性抗体,使得实验效率受限。如何在不干扰活细胞正常生理状态的前提下,精准“捕捉”这些来去匆匆的关键调控因子,一直是植物转录调控研究中的瓶颈。针对这一问题,团队巧妙融合了两种功能模块:一方面利用dCas9作为“导航系统”,可精准锚定特定DNA序列;另一方面引入TurboID酶作为高效的“标签工”,能在邻近空间内快速对蛋白质进行生物素标记。由此构建的CSPL系统,在植物细胞中表达dCas9-TurboID融合蛋白后,可靶向目标DNA区域,并对其周围蛋白进行原位标记,随后通过链霉亲和素富集与质谱分析,高效鉴定出与该序列互作的全部蛋白。
研究团队以光温响应通路中的关键基因PIF4(已知参与甘蓝叶球发育调控)为例,在结球甘蓝、拟南芥和水稻三种植物中对其启动子区域进行了系统“扫描”。结果不仅成功捕获了TCPs、FHY3等已报道的PIF4上游转录因子,以及ELFs、PWR等表观调控因子,还首次发现了GTL2、LSH3等全新的潜在调控因子。这一成果充分验证了CSPL系统在不同植物物种中的高灵敏度与广泛适用性。
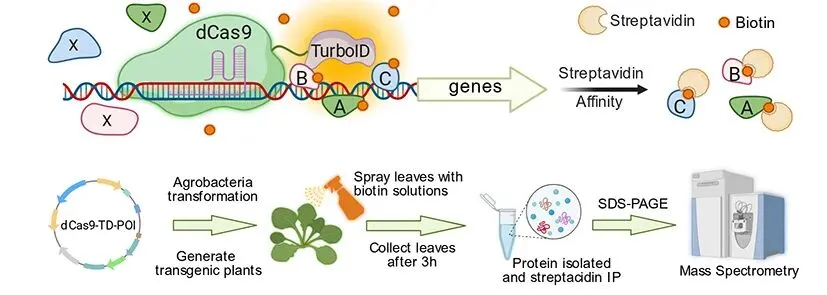
目前,CSPL技术已展现出作为通用平台的巨大潜力,能够在多种植物、不同组织及多样生长条件下,高效解析任意目标基因的上游调控网络。除PIF4外,团队正将该技术应用于甘蓝叶球驯化关键基因ACS等的调控机制研究,旨在全面揭示叶球形成背后的分子逻辑。
本论文由中国农业科学院蔬菜花卉研究所张磊副研究员、蔡成成副研究员及硕士研究生陈秋杰共同担任第一作者,程锋研究员为通讯作者。

